R İle MRI üstünde İşlemler
Bu yazı “Introduction to Neurohacking with R” dersinde1 yaptığım uygulamaları2 ve notları içerir.
DICOM formatı:
Digital Imaging and Communications in Medicine, tıbbi görüntüleme bilgileriyle çalışırken kullanılan bir standarttır. Bu çalışmadaki örnek formatımız olan MRI gibi radyolojik görüntüleme teknikleri için kullanılan temel bir formattır.
NIfTI formatı:
NIFTI, analiz stili bir veri formatıdır ve MRI verisi üzerinde inceleme yapabilmeyi sağlar. FSL, AFNI gibi bu alanda kullanılan çoğu araç bu formatı desteklemektedir. Dahası, DICOM formaından NIfTI’ye birebir çevirme şansı vardır.
İki format arasındaki farklar:
- DICOM .dcm uzantılı, NIfTI dosyaları da .nii uzantılıdır.
- DICOM dosyası görüntüyü kesitler halinde gösterirken, NIfTI dosyası sayılardan oluşan 3 boyutlu bir formattır.
- NIfTI hastaya dair bir bilgi içermezken, DICOM formatı hasta verilerini içerir.
DICOM dosyasını R ile okumak:
Hastaneler çoğunlukla DICOM formatında verileri depolamaktadır. Çalışması daha kolay olan NIfTI formatına aktarabiliriz. Bunun için ihtiyacımız olan paket:
package="oro.dicom"
ve komut:
all_slices <- readDICOMFile(filename)
DICOM -> NIFTI
nifti_file <- dicom2nifti(all_slices)
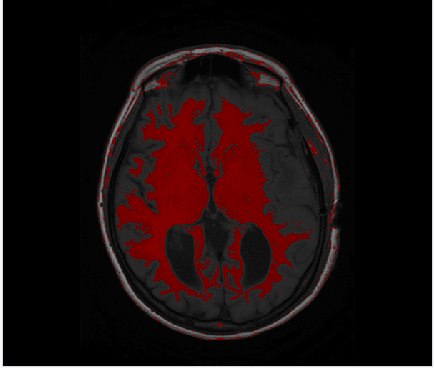
Mask nedir:
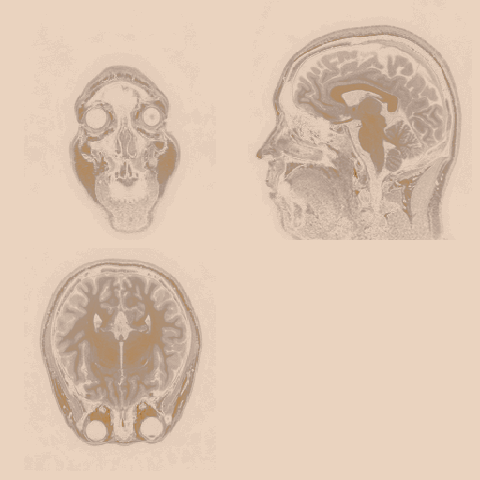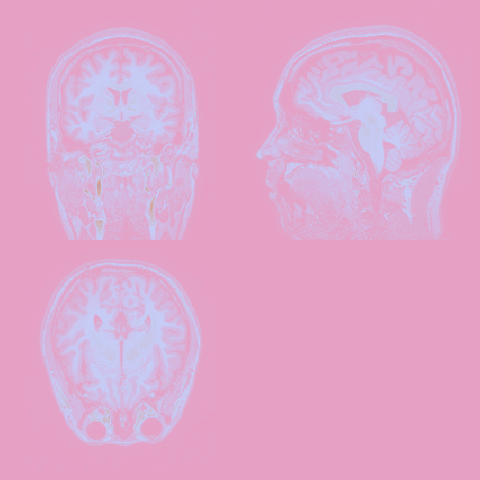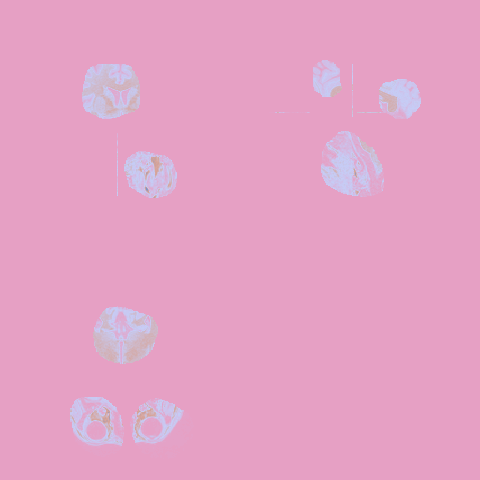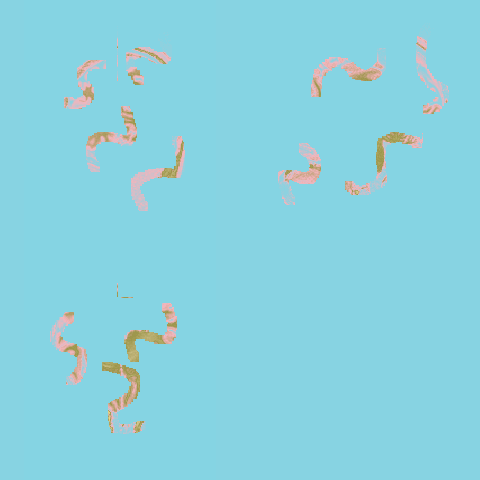
MRI verisinde odaklanmak istediğimiz noktaları öne çıkarmak amacıyla bir bölgenin veya dokunun ayrılmasına yararlar. Aşağıdaki örnekte yoğunlukları 300, 400 arasında olan dokuları yani white matterı maskelemiş oldum:
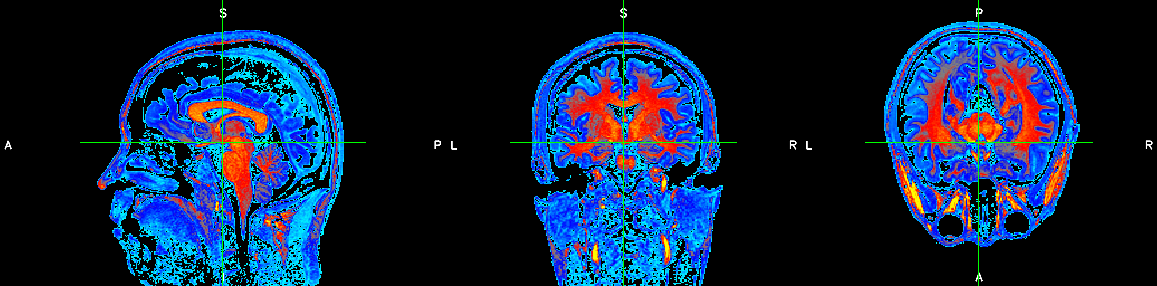
FSLeyes:
FSL aracı FMRI, MRI and DTI formatlarındaki beyin görüntülerini analiz etmek için gelişmiş bir kütüphanedir. FSLeyes programı ise FSL kullanan bir görüntüleme aracıdır. Aynı zamanda veri üstünde basit düzenlemeleri yapmaya da imkan tanıyor. Farklı parametrelerle görüntüyü renklendirmek de mümkün, amaca yönelik olarak bu da inceleyen kişiye kolaylık sağlayabilir.
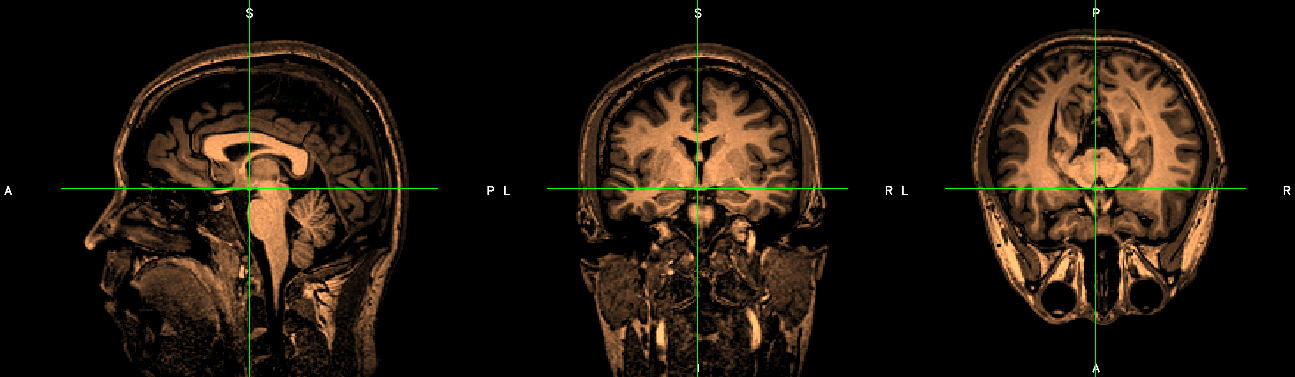
Mask oluşturma:
FSLeyes programını kullanarak masklar oluşturmayı denedim, aşağıda gördüğünüz ekran görüntüsündeki 3 ayrı beyin görüntüsü xyz eksenlerindeki aynı beynin aynı kesitinin görüntüsü. İmleçleri kullanarak beyin içinde 3 boyutlu olarak hareket etmek mümkün.
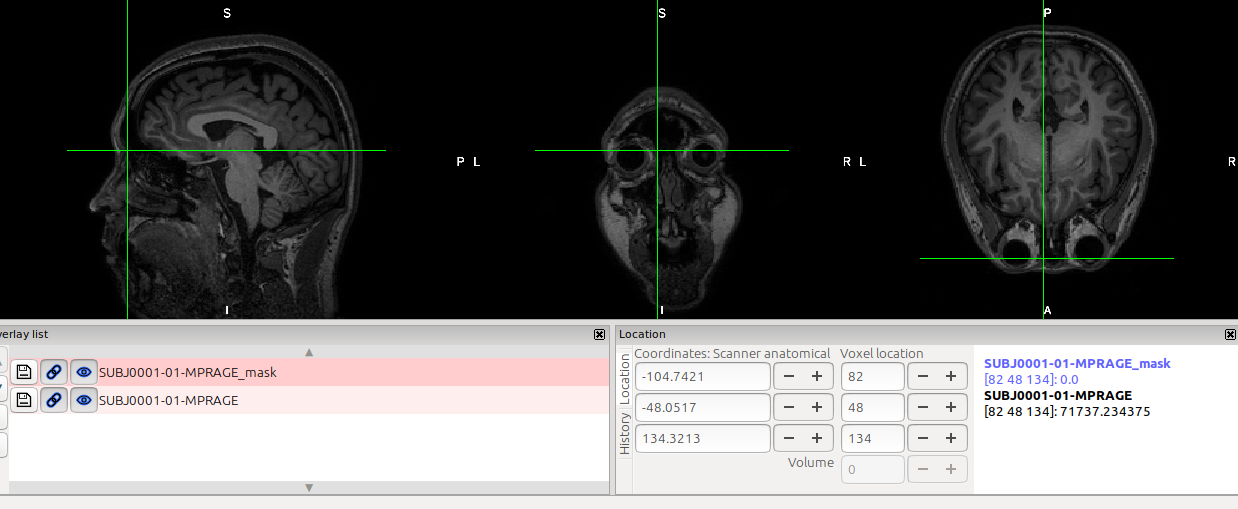
Sonuç:
Bu araçları keşfetme sürecimde ben FSLeyes programıyla bir grup beyin mask’ı oluşturdum ve NIfTI formatına çevirdim. Bunların voxel kordinatlarını R üstünde DICOM formatından NIfTI formatına çevirmiş olduğum 3 boyutlu beyin görüntüsüyle eşledim.
brainImage <- readNIfTI(file.path(mrdir,"SUBJ0001-01-MPRAGE.nii.gz"),reorient = FALSE)
mask1 <- readNIfTI(file.path(mrdir,"SUBJ0001-01-MPRAGE_mask_1.nii.gz"),reorient = FALSE)
O kordinatlar üzerinde maskeleme yaparak bazı görüntüler elde ettim. Hazırladığım örnek bir mask şu:

Görüldüğü üzre mask, aslında 1 ve 0’lardan oluşan bir 3 boyutlu sayılar arrayi. Dolayısıyla bir maskeyi aynı boyutlu bir görsel ile çarpmak demek, masklanan bölgeleri gösterip, diğer yerleri gizlemek anlamına geliyor. R üstünde maskeleme işlemi basit bir çarpma işlermi olarak yapılabiliyor.
masked1 <- brainImage*mask1
Tüm bunların sonucunda, oluşturduğum masklar ve beynin farklı açılarıyla, “wes_palette” renk paletlerini de kullanarak şöyle bir gif ortaya çıkardım:
Koda buradan ulaşabilirsiniz.